Les archéobactéries seraient les ancêtres
des plantes, des animaux… et de l’Homme!
Par W. Ford Doolittle
Nous, les humains, sommes par nature des classificateurs. Nous cherchons toujours, dans le monde qui nous entoure, des similitudes. Si nous ne les trouvions pas, nous serions contraints de considérer chaque nouveau rocher, chaque nouvelle rivière, chaque nouvel arbre, comme un phénomène inconnu à propos duquel il nous serait impossible de faire aucune prédiction raisonnable. Nous serions vite submergés par la complexité du monde naturel.
Notre goût des classifications (ou taxonomies, du grec taxis, arrangement, ordre, et nomos, administration), nous l’avons plus particulièrement développé dans le domaine de la vie. Les sociétés humaines attribuent en effet des noms d’espèces aux organismes vivants qu’elles trouvent sur leur chemin ou qu’elles récoltent pour survivre, et ont mis au point des méthodes pour intégrer intellectuellement les organismes vivants dans desensembles conceptuels de plus en plus larges (que l’on appelle aussi taxons), comme c’est par exemple le cas dans la proposition: «Les rouge-gorges et les roitelets sont des oiseaux, les ours et les lions sont des mammifères, les oiseaux et les mammifères sont des animaux».
Le fait de pouvoir distinguer les plantes et les animaux comestibles des plantes et des animaux toxiques a d’évidence permis à l’espèce humaine de survivre au cours de l’évolution. Même si ce genre de connaissances se transmet par la culture, on ne peut se défaire de l’impression, néanmoins, que la prédisposition à classer les choses est un comportement inné de l’homme, littéralement enfiché dans son cerveau.
C’est sans doute la raison pour laquelle les biologistes modernes éprouvent une inextinguible passion pour les classifications, même s’ils ne sont plus contraints de collecter leur nourriture dans la jungle ou de chasser le tigre pour survivre. Cette passion est particulièrement vive chez les biologistes spécialisés dans l’étude de l’évolution des espèces. Ils rêvent en effet de reconstituer (intellectuellement, bien sûr) le processus d’évolution de la vie au cours des 4 derniers milliards d’années, et d’expliquer comment pressions et contre-pressions ont abouti au monde vivant incroyablement divers que nous connaissons aujourd’hui. Mais s’ils veulent avoir une chance de réussir, il leur faut absolument connaître les embranchements exacts de l’arbre de la vie, c’est-à-dire, en définitive, les relations de parenté exactes existant ou ayant existé entre les espèces. Bref, ils doivent savoir qui descend de qui!
La quête n’est pas purement gratuite. Si l’on ignore de quelle manière les organismes vivants actuels (de l’observation desquels nous tirons l’entier de nos connaissances en matière de physiologie, de biochimie et de génétique) sont reliés les uns aux autres, il s’avère en effet impossible de dire quoi que ce soit d’utile sur les origines de la photosynthèse, sur la première apparition des organismes multicellulaires, sur la diversification des vertébrés, sur l’évolution de l’intelligence chez les primates, pour ne mentionner que quelques exemples.
Avant que n’existent les microscopes, on disait ordinairement que toutes les formes de vie appartiennent à l’un de deux grands ensembles majeurs: le règne animal (Animalia) et le règne végétal (Plantae). Mais l’invention des premiers microscopes fit découvrir aux savants du XVIIe siècle une myriade d’organismes qui entraient mal dans ces catégories: les algues et champignons unicellulaires, les protozoaires, les bactéries. Comme ces organismes se ressemblaient par leur petite taille et leur apparente simplicité, ils se contentèrent de les fourrer en vrac dans un troisième règne, imaginé pour la circonstance, les Protista.
Dans les années 1930 et 1940, l’invention et le développement du microscope électronique fit faire un bond à notre compréhension du monde vivant et nous obligea à modifier les bases mêmes de notre système de classification. Il apparut en effet que les animaux, les plantes, les champignons et les «protistes supérieurs» (algues et protozoaires), classés jusque là dans des règnes différents, se ressemblaient néanmoins sur un point essentiel, à savoir que tous sont constitués de cellules dans lesquelles le matériel génétique (le fameux ADN) est enfermé dans un noyau , lequel est donc séparé du reste de la cellule (le cytoplasme) par une membrane. On nomma donc ces organismes des eucaryotes (ou vrai noyau), alors que l’on nommait par contraste procaryotes (ou avant noyau) les organismes — les bactéries, les algues bleu-vertes, les cyanobactéries, etc. — dont le matériel génétique flotte librement dans le cytoplasme.
En d’autres termes, on décida de répartir toutes les formes de vie connues dans deux «super-règnes», lesEukaryota et les Prokaryota, qui, durant les années 1950, 60 et 70, dominèrent de la tête et des épaules la réflexion et les expériences de tous les biologistes de la Terre. A cette époque, tous étaient pratiquement d’accord avec Roger Stanier, un biologiste canadien, qui disait que le principal embranchement dans l’évolution du vivant s’est produit le jour où les organismes cellulaires eucaryotiques et procaryotiques se sont séparés.
Il y a deux manières possibles de classifier les organismes vivants. On peut considérer leur apparence physique et leur comportement, et mettre par exemple toutes les créatures qui se servent d’ailes pour voler dans un groupe, tous les animaux qui ont des écailles et le sang froid dans un autre, et ainsi de suite. Mais on peut aussi recourir à la généalogie, et mettre dans une même catégorie les espèces les plus récemment dérivées d’un ancêtre commun, même si ces espèces dérivées ont des comportements et des apparences complètement divergents (généalogiquement, les oiseaux et les crocodiles, qui ont l’air vraiment très différents, sont plus proches les uns des autres que les crocodiles et les lézards).
La science, comme d’ailleurs le bon sens, soutiennent qu’il ne peut y avoir de parenté que généalogique. Raison pour laquelle si l’Homme réussissait un jour à créer de toutes pièces la copie chimiquement parfaite d’une souris, cette dernière ne pourrait, par définition, appartenir au genre Mus (les muridés) puisqu’elle ne serait pas descendue de l’ancêtre commun du genre.
Hélas, il est rarement possible d’observer le développement d’une longue lignée généalogique. C’est pourquoi les biologistes ont pris l’habitude de déduire de ressemblances comportementales, morphologiques, physiologiques et biochimiques l’existence d’une parenté entre espèces vivantes (ou entre espèces vivantes et espèces fossiles). A ce jeu des déductions, on peut dire qu’ils n’ont pas mal réussi, et déjoué la plupart des pièges. Ils ont su éviter, par exemple, de classer dans un même groupe les oiseaux et les chauves-souris, bien qu’ils aient développé les uns et les autres des ailes pour voler, parce qu’ils ont eu la sagesse de considérer d’autres critères, notamment que les chauves-souris, à la différence des oiseaux, sont vivipares et possèdent des mamelles.
Or depuis le milieu des années 60, les chercheurs disposent enfin d’outils de classification vraimentgénéalogiques, fondés directement sur la séquence d’ADN d’une cellule, ou sur les séquences d’ARN et de protéines copiées de l’ADN. L’idée de base est simple. imaginez qze d’une espèce ancestrale X découlent par mutations deux nouvelles espèces A et B, et que de ces espèces découlent plus tard A’ et A » et B’ et B ». Pratiquement cela se traduira par le fait que tel morceau d’un gène de X ayant, disons, la séquence AAATTCGTGGA deviendra le gène d’une nouvelle espèce A par mutation CAATTCGAGGA par exemple, et d’une espèce B par mutation AAATTCGTGAC (j’ai noté les mutations en gras) produiront les nouvelles espèces A’ et A », mais malgré ces chamboulements, leurs séquences se ressembleront davantage entre elles que les séquences correspondantes de B’ et B ». [Rappelons ici que l’ADN est fait de deux rubans tournoyant en hélice, chaque ruban portant une succession de « bases » reliées chimiques aux bases de l’autre ruban. Les lettres A, T, G et C sont les abréviations des quatre bases (adénine, thymine, guanine et cytosine), structures chimiques dont est constitué l’alphabet du code génétique].
Si, comme cela est généralement le cas, le biologiste ne dispose que des séquences des espèces existant aujourd’hui (disons les séquences de A’, A », B’ et B »), il peut néanmoins, en comparant habilement ces séquences et en raisonnant en arrière, si je puis dire, déduire de quelle manière ces espèces sont parentes, et comment elles se relient aux espèces intermédiaires éteintes A et B, et à l’ancêtre commun X. Il est vrai que là, de nouveau, le biologiste est obligé de raisonner par déduction, mais au moins travaille-t-ildirectement sur de l’information héréditaire. Compte tenu du fait que tout changement durable de la biologie d’une espèce s’inscrit dans son ADN, on peut dire que les séquences d’ADN sont au biologiste ce que les registres paroissiaux et civils enregistrant mariages, naissances et décès sont aux spécialistes des généalogies humaines.
Emile Zuckerkandl et Linus Pauling ont, en 1965, suggéré les premiers qu’on pourrait reconstituer un jour la totalité de l’histoire de la vie à partie des séquences des bases dans le matériel génétique de diverses espèces. Mais avant que leur intuition ne se vérifie dans les faits, il fallut attendre la révolution biotechnologique des années 70 et 80, qui permit non seulement d’analyser les séquences d’ADN, mais surtout de recombiner et cloner (c’est-à-dire reproduire à l’identique) les éléments de matériel génétique mis ainsi en évidence, et avant qu’elle ne débouche sur une remise en cause de la classification des espèces, que Carl Woese prenne la peine de combiner les outils révolutionnaires de la biotechnologie avec les idées de Zuckerkandl et de Pauling.
Woese, professeur à l’Université de l’Illinois, à Urbana, entreprit en effet de classer tous les organismes vivants en utilisant la séquence d’une molécule que produisent toutes les cellules, l’ARN ribosomal «16S». Cette molécule étant essentielle à la lecture du code génétique, elle a peu changé depuis l’apparition des premières cellules, il y plus de 3,5 milliards d’années, mais suffisamment néanmoins pour que les différences obervées entre les séquences de quelque 1500 bases dans l’ARN de 16S de différents organismes permettent de reconstruire l’évolution des espèces depuis les racines de l’arbre de la vie.
A l’époque où Woese avait entrepris d’analyser les différentes séquences des ARN de 16S, les outils pour le faire étaient encore primitifs et les perspectives de succès incertaines. On devine donc la surprise de ses collègues lorsqu’il leur annonça, en 1977, qu’il avait découvert «une nouvelle forme de vie», les archéobactéries (archaebacteria). Ses analyses de 16S l’avaient en effet convaincu que les bactéries méthanogènes, qui vivent dans les étangs, dans le rumen [matière ruminée] des vaches, dans l’intestin des termites, et qui sont responsables de la production biologique de méthane, diffèrent complètement de la majorité des autres procaryotes, qu’il rebaptisa eubactéries (rappelez-vous: les procaryotes sont des cellules sans noyau).
Les années suivantes, sur la base de nouvelles analyses de 16S, il annonça coup sur coup qu’il avait découvert deux autres groupes d’organismes sans noyau faisant partie, à son sens, du nouveau groupe des archéobactéries: les halophiles extrêmes, qui ont besoin de très hautes concentrations de sel pour se développer, et les thermophiles extrêmes, qui ont besoin de très hautes températures pour vivre.
Ses confrères, inquiets, commencèrent à se demander s’il était vraiment possible de reconstruire la totalité de l’évolution des espèces à partir du seul 16S. De nouvelles recherches apaisèrent cependant leurs doutes. Et aujourd’hui, plus personne ne doute que les archéobactéries, dont la paroi cellulaire, la biologie moléculaire, la biochimie, sont différentes de celles des autres procaryotes, forment un groupe distinct et cohérent. Oui, dans l’univers biologique actuel, il existe bien deux sortes de procaryotes (ou cellules sans noyau) très différentes.
Il est vrai que si les archéobactéries se ressemblent au niveau cellulaire, leurs capacités d’adaptation au milieu diffèrent considérablement.
Certaines bactéries thermophiles peuvent supporter des températures dépassant de beaucoup 100 degrés Centigrade. Les températures les plus élevées se produisent sous haute pression, autour des échappements d’eau chaude sous-marins par exemple, où prolifère une faune archéobactérienne. Chaque molécule de protéine des bactéries thermophiles extrêmes doit non seulement être capable conserver sa stabilité à des températures qui détruiraient toute protéine humaine, mais aussi de fonctionner à ces températures de manière optimale. Comme bien l’on imagine, les chercheurs travaillent aujourd’hui comme des déments afin de découvrir le secret des structures qui donnent une telle stabilité à ces protéines, et d’imaginer à cette propriété extraordinaire des usages industriels.
Les protéines des bactéries halophiles, quant à elles, sont hautement résistantes au sel. C’est ce qui explique qu’elles se développent joyeusement sur le poisson salé; elles y produisent un pigment rouge qui a donné son nom au red herring, hareng «rouge» ou hareng saur.
Les protéines des bactéries méthanogènes, enfin, meurent empoisonnées lorsqu’on les oxygène, parce que leur environnement habituel est privé d’air.
Toutes les archéobactéries ne vivent cependant pas en milieu extrême. Plusieurs indices permettent de penser qu’elles constituent une proportion importante de la flore microbienne des océans.
Revenons cependant à notre problème de classification des espèces. Puisqu’il y a désormais deux types de procaryotes (deux types de cellules sans noyau), on est en droit de se demander quels sont les liens de parenté de ces cellules sans noyau avec les eucaryotes (ou cellules avec noyau)? Quels sont les ancêtres, quels sont les descendants?
Pendant une dizaine d’années, il a semblé impossible de répondre à cette question de l’œuf et de la poule. Mais en 1989, une équipe japonaise emmenée par Takashi Miyata, mit au point une méthode astucieuse, fondée sur l’analyse des séquences de gènes très anciens, et démontra que les deux premières branches de l’arbre de la vie (juste au-dessus des racines, c’est-à-dire juste après le début de la vie) sont les eubactéries et les archéobactéries, et que les eucaryotes ne sont qu’un embranchement ultérieur des archéobactéries.
Cette découverte bouleversa les biologistes presque autant que le découverte originelle des archéobactéries douze années plus tôt. Jusque là, en effet, la plupart de mes collègues et moi-même éprouvions le sentiment diffus que les archéobactéries n’avaient au fond rien de commun avec les eucaryotes (vrai noyau), précisément parce qu’elles n’avaient pas de noyau, et qu’elles n’avaient pas non plus les structures cellulaires complexes des eucaryotes… Et aussi parce que ceux qui cultivent et manipulent les archéobactéries sont des bactériologistes, pas des botanistes, ni des zoologues. Or voici que l’on nous disait, tout à trac, que ces minables archéobactéries étaient les ancêtres des eucaryotes — c’est-à-dire des plantes, des animaux, de l’Homme!
Le choc fut tel qu’une résistance surgit le jour où Woese suggéra d’abandonner complètement la grande division binaire procaryotes/eucaryotes, qui avait si bien servi la biologie durant ce siècle, pour la remplacer par une division en trois super-règnes de rang taxonomique égal: les eubactéries (Bacteria), les archéobactéries (Archaea) et les eucaryotes (Eukarya). La résistance vint non seulement de bactériologistes arguant que cette réorganisation allait en pratique semer la pagaille, mais aussi de théoriciens de l’évolution comme Ernst Mayr, qui considéraient que l’apparition de la très complexe cellule eucaryotique était le deuxième pas le plus radical et le plus extraordinaire de l’évolution de la vie sur Terre (le premier ayant été évidemment l’apparition de la vie elle-même), et qu’il était donc exclu d’en minorer l’importance en instaurant une nouvelle classification.
C’est à ce moment qu’entrèrent dans le bal les biologistes moléculaires, qui, avec leurs instruments d’analyse puissants, avaient entrepris d’étudier la «transcription», qui est l’un des mécanismes les plus fondamentaux de la vie. On appelle transcription le processus par lequel l’information héréditaire (transmise de génération en génération sous forme d’ADN) se diffuse pratiquement, jour après jour, dans la vie de la cellule et de l’organisme. La transcription est représentée par la première flèche dans l’expression classique du «dogme central de la biologie moléculaire»: ADN —> ARN —> protéine. En d’autres termes, le produit de la transcription est l’ARN (ou plus exactement un «ARN messager»), dont la séquence est, à part une légère modification chimique, la copie exacte d’un fragment d’ADN comprenant un gène et correspondant à une protéine cellulaire unique.
Comme les cellules ont pratiqué l’art de la transcription depuis avant même l’époque du «dernier ancêtre commun», c’est-à-dire de la cellule primitive qui a donné naissance tant aux eubactéries qu’aux archéobactéries et aux eucaryotes, aujourd’hui encore, 3,5 milliards d’années plus tard, de fortes similitudes demeurent dans la manière dont les cellules de ces trois super-règnes pratiquent leur transcription.
Mais il y a aussi des différences cruciales.
Chez les eubactéries, une enzyme relativement simple — l’ARN-polymérase — transcrit l’information qui était codée dans l’ADN directement dans l’ARN.
Chez les eucaryotes, les choses sont plus compliquées. L’ARN-polymérase y est constituée de beaucoup plus de composants protéiques, y est beaucoup plus massive et complexe, et n’y entre pas en contact direct avec l’ADN. Elle a besoin en effet d’autres protéines, appelées «facteurs de transcription», qui reconnaissent les régions proches des gènes à transcrire, s’y fixent et, telles des sirènes, attirent les complexes d’ARN-polymérases et les encouragent à rendre visite aux gènes proches et à les transcrire. En fait, la plupart des gènes eucaryotiques ont besoin, pour pouvoir être transcrits, de plusieurs «facteurs de transcription», eux-mêmes liés souvent à d’autres protéines encore — au point d’ailleurs que la biologie moléculaire eucaryotique actuelle passe le plus clair de son temps à énumérer ces protéines additionnelles et à leur trouver des noms!
Quant aux archéobactéries, enfin, Wolfram Zillig et ses collègues, à Munich, ont montré, dans les années 80, que leurs ARN-polymérases ressemblent plus à celles des eucaryotes qu’à celles des eubactéries: elles sont en effet grandes, et constituées de nombreuses protéines elles-mêmes liées à des composants supplémentaires. Des essais entrepris à cette époque pour réaliser en laboratoire une transcription archéobactérienne échouèrent. Certains facteurs manquaient.
Quels facteurs? Nous commençons tout juste à découvrir qu’il s’agit de diverses protéines, clairement parentes des «facteurs de transcription» eucaryotiques, à telle enseigne qu’en laboratoire on peut parfois remplacer avec succès les protéines archéobactériennes par des «facteurs de transcription» eucaryotiques. Sur ce point fondamental de la vie de toute cellule, les archéobactéries ressemblent donc indubitablement aux eucaryotes.
Le tableau final qui résultera de cette formidable enquête à la Sherlock Holmes sera, à mon avis, à peu près le suivant.
On verra que certains éléments de base de la machinerie génétique des archéobactéries et des eubactéries se ressemblent beaucoup, que leurs chromosomes notamment s’organisent et se dédoublent de manière presque identique. On en déduira qu’il s’agit là de l’organisation «primitive» de la vie, très proche de l’organisation de la cellule ancestrale commune. Mais on verra aussi que plusieurs systèmes d’expression et de régulation des gènes, qui ont atteint chez les eucaryotes un degré d’élaboration et de complexité incroyablement élevé, ont déjà commencé à se compliquer chez les archéobactéries. On verra également que si l’enfermement du noyau et la réorganisation de la cellule qui ont marqué la naissance des premiers eucaryotes ont été dans l’évolution de la vie des événements exceptionnels, d’autres traits que l’on croyait uniques aux eucaryotes étaient connus déjà des archéobactéries. Du coup, la ligne de démarcation entre les eucaryotes et les procaryotes perdra de sa netteté, et les archéobactéries apparaîtront alors, en effet, comme le «chaînon manquant», ainsi que les avaient surnommées, au moment de leur découverte, en 1977, des journalistes enthousiastes.
Ce ne sont là, bien sûr, que des prédictions. Mais nous en aurons bientôt le cœur net. Nous disposerons bientôt d’une profusion de données. La communauté biomédicale mondiale se prépare à analyser les séquences des quatre milliards de bases du génome humain. Cependant que plusieurs laboratoires publics et privés, au Canada, aux États-Unis et en Europe, avancent à grands pas dans l’analyse des séquences de plusieurs génomes procaryotiques — trois millions de bases environ — pour se faire la main avant le grand séquençage du génome humain, pour essayer de trouver des gènes impliqués dans le développement de certaines maladies humaines, pour tenter de trouver de nouvelles protéines à usage industriel, et, last but not least, pour faire avancer les connaissances pures de la biologie de l’évolution… dont les serviteurs, le lecteur s’en sera convaincu j’espère, ont encore de beaux jours devant eux.
© Le Temps stratégique, No 67, décembre 1995.
ADDENDA
La grande foire aux gènes
Étape No 1: visite guidée de l’ADN
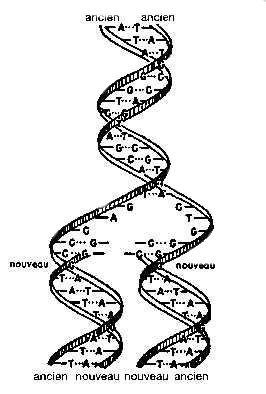
L’ADN (ou acide désoxyribonucléique) est le matériel où est stockée l’information génétique et l’ensemble des caractères héréditaires d’une cellule. Sa structure moléculaire consiste en une sorte d’échelle constituée de quatre sortes de barreaux; elle est représentée par une double chaîne de nucléotides de forme hélicoïdale. Les quatre nucléotides, sous-unités de l’ADN, contiennent les bases azotées adénine (A), guanine (G), cytosine (C) et thymine (T). Les bases s’assemblent selon une complémentarité exclusive: A s’apparie uniquement avec T, et G avec C.
Le rôle fondamental de l’ADN est de stocker l’information génétique. L’ADN est le matériel (« hardware ») dépositaire des caractères héréditaires, la mémoire du code génétique des êtres vivants. Le matériel génétique de l’ADN peut servir à être reproduit tel quel (lors de la réplication) ou traité en vue d’élaborer de nouvelle molécules nécessaires au métabolisme des cellules (durant les opérations de transcription et de traduction).
L’ARN, qui du point de vue de sa structure moléculaire est similaire à l’ADN, se distingue par son rôle essentiel de messager de l’information génétique. L’ARN est un intermédiaire-convoyeur entre l’ADN (dont il copie « en négatif » une séquence d’information) et les structures cellulaires, chargées de lire la séquence d’information copiée de l’ADN en vue de la production des protéines. Il est en quelque sorte le « logiciel » de la cellule. Il existe différents types d’ARN (ARN messager, ARN de transfert, ARN ribosomal) qui tous ont un rôle particulier dans le processus complexe de synthèse des protéines.
[titre figure 1:] Modèle structural de l’ADN
[figure 1, source. Jacques Mauel]
[Informations:]
Phosphate
Sucre
distance entre deux couples de bases 3.4 Å
pas de l’hélice 34 Å
Etape No 2: visite guidée de l’ARN
L’ARN (ou acide ribonuléique) est une susbstance chimique existant dans le cytoplasme, et le cas échéant dans le noyau, cellulaires. L’ARN est le messager de l’information génétique codée dans l’ADN, il joue un rôle majeur dans les opérations de transcription et de traduction de l’information codée dans l’ADN.
Il existe différents types de molécules d’ARN qui assurent chacune une fonction particulière dans la messagerie de l’information génétique.
L’ARN messager (ARN m), se forme au contact de l’ADN et son rôle consiste à transcrire une séquence d’ADN puis de transporter l’information génétique recueillie du noyau vers le cytoplasme. L’ARN messager va ensuite se placer sur une unité d’assemblage des protéines, le ribosome, où il sera traduit pour élaborer une séquence d’acides aminés nécessaires à la synthèse des protéines.
L’ARN ribosomal représente 80 % de l’ARN total d’une cellule. Associé à des protéines, il forme le ribosome qui constitue la tête de lecture de l’information génétique transcrite par l’ARN messager. C’est dans le ribosome que sont enchaînées les séquences d’acides aminés qui constituent les molécules de protéine. Une des formes de l’ARN ribosomal est la fameuse séquence 16 S mentionée dans le texte de W. Ford Doolittle.
Les ARN de transfert (ARN t) sont des molécules qui se placent sur les sites du ribosome où va être lu l’ARN messager. Leur rôle fondamental est celui d’adaptateurs car ils permettent de reconnaître les acides aminés dans le cytoplasme pour les amener jusqu’au brin d’ARN messager et les positionner de manière à ce que leurs enchaînement dans la protéine à synthétiser corresponde aux instructions précisées par la séquence des bases du segment d’ADN codant pour cette protéine.
Ces deux formes d’ARN (ARN ribosomal et ARN t) interviennent dans la phase de traduction, où il s’agit de décrypter ou traduire l’information génétique convoyée par l’ARN messager.

Comment recopier de l’ADN
La réplication ou duplication correspond au processus de synthèse de l’ADN. Cette opération intervient au cours de la division cellulaire pour transmettre aux cellules filles les caractères héréditaires de la cellule mère.
La duplication commence lorsque les deux brins de la double hélice d’ADN s’ouvrent, à la façon d’une fermeture éclair. Chaque brin isolé va ensuite servir de matrice à un brin complémentaire qui vient s’imbriquer selon la complémentarité des bases (A ,T, G, C). Deux paires de chaînes sont ainsi formées, identiques à la chaîne mère.
Y a-t-il une vie sous la vie
cachée dans les entrailles de la Terre?
Il existe, très profond dans la Terre, de nouvelles formes de vie microbienne dont l’abondance est telle, que certains scientifiques commencent à se demander si notre planète ne cache pas dans ses entrailles une biosphère de masse totale égale ou supérieure à celle de la biosphère de surface. Si une telle biosphère profonde existait , disent ces scientifiques, il faudrait récrire nos théories sur les origines de la vie. Beaucoup de leurs collègues affichent cependant leur scepticisme, même s’ils trouvent la thèse suffisamment intrigante pour que l’on multiplie les études sur le monde souterrain.
Des microbes qui se nourrissent de la chaleur de la Terre
Ce qui est extraordinaires chez ces « nouveaux » microbes est qu’ils se développent à des températures mortelles pour toutes les autres formes de vie: 110 degrés Centigrade, par exemple, voire 370 degrés Centigrade pour des périodes brèves. Alors que la plupart des microbes terrestres meurent avant que la température n’atteigne100 degrés Centigrade.
On a découvert de ces microbes hyperthermophiles dans des sources chaudes, des cratères de volcans en activité, des conduits sous le fond des océans, et, plus récemment, dans des nappes de pétrole profondes. Certains scientifiques disent que ces microbes pourraient être omniprésents dans les premiers kilomètres de la croûte terrestre, tapis dans les pores emplis de fluides, dans les craquelures et les interstices des roches, se nourrissant d’éléments chimiques souterrains et se réchauffant à la chaleur de la Terre. Leur habitat principal se situerait cependant sous les continents et dans les fosses océaniques, où ils se nourriraient d’aliments charriés par les flux lents de pétrole et d’eaux profondes. Ils fonctionneraient grâce à l’énergie interne de la Terre, par opposition aux organismes de surface, qui reçoivent généralement leur énergie de la lumière du Soleil, par le biais de la photosynthèse.
Bien à l’abri des tempêtes de surface
Pour Norman R. Pace, biologiste à l’Université de l’Indiana, « il y a sous nos pieds, potentiellement, une énorme quantité de biomasse. Mais l’on ignore quelle est sa profondeur, sa température, et combien il y en a. En tout cas, chaque fois que les conditions chimiques sont adéquates, la vie surgit. »
John A. Baross, biologiste à l’Université de Washington, à Seattle, pense lui aussi que la biosphère profonde existe et même qu’elle est le lieu où est née la vie, parce qu’il se trouvait à l’abri des bombardements de météorites et des radiations qui, à l’époque où la Terre s’est formée, rendaient sa surface absolument invivable.
La découverte d’une biosphère profonde, disent les scientifiques, suggérerait que des agents biologiques ont participé à la formation du pétrole et du gaz, et contribué à concentrer dans la croûte terrestre des métaux rares tels que l’or, l’argent et le platine. La biosphère profonde, si elle existe, pourrait être par ailleurs, pour l’industrie biotechnologique, une réserve inépuisable de biodiversité génétique.
Oui, mais reste à savoir si c’est vrai.
Certains scientifiques tempèrent cependant cet enthousiasme. Holger W. Jannasch, microbiologiste expérimental à la Woods Hole Oceanographic Institution, à Cape Cod, dans le Massachusetts estime par exemple que l’on ne dispose pas d’observations assez nombreuses pour pouvoir affirmer qu’une biosphère profonde existe. Il faudrait, pour que j’en sois convaincu, dit-il, que les microbes souterrains soient capables de supporter des températures bien plus élevées encore que celles qu’ils semblent aujourd’hui capables de supporter.
William J. Broad, © New York Times Service.